Искусственный интеллект рассчитал 3D-структуры сотен белковых комплексов
С помощью программ RoseTTAFold и AlphaFold ученые проанализировали 8,3 млн. пар белков дрожжей, определили среди них те, которые образуют комплексы, и для многих комплексов вычислили структуру с высоким разрешением. Это очередной крупный успех компьютерной структурной биологии, открывающий путь к новому уровню понимания работы клетки.
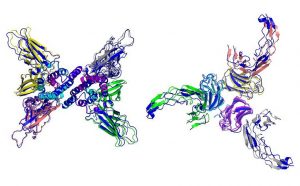
Структуры гетеромеров, вычисленные с помощью AlphaFold-Multimer
В прошлом году две научные группы, одна из британской компании DeepMind, а другая во главе с Дэвидом Бейкером из Вашингтонского университета в Сиэтле, создали программы AlphaFold и RoseTTAFold, основанные на машинном обучении. Эти программы способны с высокой точностью предсказывать трехмерную структуру белка по последовательности его аминокислот. С их помощью построены тысячи белковых структур. RoseTTAFold также позволила определить структуры нескольких известных белковых комплексов, но в основном бактериальных. Предсказание белковых взаимодействий у эукариот до сих пор казалась достаточно сложной задачей, однако последние версии программ могут с ней справиться. В новом исследовании ученые использовали комбинацию RoseTTAFold и AlphaFold, чтобы проанализировать более 8 миллионов пар белков дрожжей Saccharomyces cerevisiae и определить потенциальные взаимодействия между ними.

В исследовании участвовали сотрудники различных научных центров США и других стран, одним из руководителей был Дэвид Бейкер. Чтобы найти белки, которые могут образовывать комплексы друг с другом, команда сначала сравнила аминокислотные последовательности всех 6000 дрожжевых белков с белками других видов: 2026 грибов и 4325 других эукариот. Сравнения позволили отследить, как эти белки эволюционировали, и идентифицировать участки, которые, по-видимому, изменялись параллельно в разных белках. Исследователи предположили, что белки, образующие комплексы, «стремятся» сохранить свои взаимодействия в ходе эволюции.
Затем команда использовала программу RoseTTAFold вместе с общедоступной AlphaFold от DeepMind, чтобы попытаться построить 3D-структуры каждой пары кандидатов. Из 8,3 миллиона пар дрожжевых белков, которые предположительно коэволюционируют, программы ИИ идентифицировали 1505 вероятных белок-белковых взаимодействий. Для 699 из них имелась информация по структуре, для 700 — экспериментальные данные, и еще 106 не были описаны нигде. Модели удалось построить 3D-структуру с высоким разрешением для 806 комплексов, для которых ранее такое разрешение не было доступно. Некоторые комплексы включали до 5 субъединиц.

«Эти взаимодействия охватывают все процессы, происходящие в эукариотических клетках», — сказал соруководитель работы Цянь Конг, эксперт по биомедицинской информатике Юго-западного медицинского центра Техасского университета. Обнаруженные комплексы участвуют в репарации ДНК, митозе и мейозе, транскрипции и трансляции, транслокации и транспорте белков, метаболизме, входят в состав цитоскелета и клеточных органелл.
Компьютерные модели не заменяют данных «мокрой» биологии, но дают необходимые гипотезы для экспериментальной проверки экспериментаторами. Картирование белков до атомного масштаба требует таких дорогостоящих методов, как рентгеновская кристаллография и ЯМР-спектроскопия, а «подсказки» от ИИ могут существенно ускорить и удешевить исследование.

Действие многих лекарств основано на нарушении взаимодействий между белками, поэтому исследование белковых комплексов предоставляет разработчикам терапевтических препаратов много потенциальных мишеней.
Вероятно, в ближайшие годы структурная биология продолжит стремительно развиваться. В октябре разработчики AlphaFold разместили на сервере bioRxiv препринт, описывающий новую версию программы AlphaFold-Multimer, которая картировала структуры 4433 белковых комплексов. Анализ достоверности структур позволяет предположить, что точность составляет примерно 69%.
Цитируется по:
Ian R. Humphreys et al. Computed structures of core eukaryotic protein complexes. // Science (2021). DOI: 10.1126/science.abm4805.
Источник: https://pcr.news
18.11.2021