Новый ингибитор репликации коронавируса связывает трехмерные структуры его РНК
Большая часть сегодняшних исследований коронавируса ориентирована на поиск ингибиторов вирусных белков, таких как РНК-зависимая РНК-полимераза.
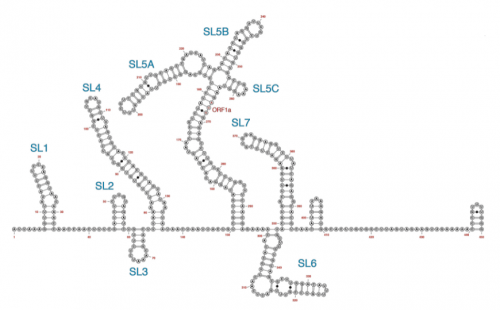
В этих случаях ингибиторы имеют дело с кодирующими последовательностями неструктурных белков, обеспечивающих размножение вируса. Эти белки более консервативны, чем структурные белки, но тоже достаточно быстро накапливают мутации. Согласно данным из геномных баз, в нетранслируемых участках генома коронавирусов мутации появляются реже. Поэтому эти области могут представлять интерес в качестве мишеней для лекарственной терапии. Ранее в 5’- и 3’-нетранслируемых областях (UTR) различных коронавирусов были идентифицированы консервативные элементы — шпилечные структуры, регулирующие вирусную репликацию. Малые молекулы, связывающие подобные структуры, были эффективны при терапии некоторых (+)-РНК-вирусов, например, вируса гепатита С. Были идентифицированы производные диметиламилорида (DMA), которые таргетируют сайт внутренней инициации трансляции (IRES) энтеровируса 71 и ингибируют его трансляцию и репликацию. В новой работе авторы исследовали возможность соединений DMA связываться со шпилечными структурами в составе 5’-UTR геномов бетакоронавирусов (к которым относятся SARS-CoV, SARS-CoV-2, MERS и простудный вирус ОС43) и ингибировать их размножение. Антивирусную активность 23 кандидатов анализировали на клеточной культуре Vero E6 и простудном человеческом коронавирусе OC43. Дозозависимое снижение вирусных титров было показано после обработки клеток тремя препаратами –– DMA-132, DMA-135 и DMA-155. Эти три соединения также значительно снижали вирусные титры в клетках, зараженных SARS-CoV-2. Для исследования механизма действия препаратов авторы сконструировали плазмиду, содержащую 805 нуклеотидов с 5’-конца генома SARS-CoV-2 (5’-UTR и часть кодирующей последовательности), 3’-UTR и репортерный ген (люциферазу). РНК с такой плазмиды трансфицировали клетки, которые затем обрабатывали различными концентрациями соединений DMA. Обработка препаратами DMA-132 и DMA-135 снижала уровень экспрессии гена-репортера слабее, чем обработка DMA-155. Удаление из конструкции плазмиды участка 3’UTR не влияло на результаты, из чего следует, что действие DMA-препаратов нацелено на 5’-участок. Взаимодействие DMA со шпилечными структурами исследовали методом TROSY HSQC (гетероядерная одноквантовая корреляционная спектроскопия). Для моделирования трехмерных структур исследуемых участков использовали программу FARFAR, с помощью которой сгенерировали 5000–100 000 моделей для каждой шпильки. Наиболее подходящие конформации исследовали с помощью программы ICM pocket finder, которая находит в трехмерных структурах области связывания различных лигандов. Полученные результаты позволили идентифицировать на 5’-концевом участке РНК четыре шпильки, обладающие наиболее сильным потенциалом для формирования комплексов с DMA-препаратами. Оптимальной мишенью для связывания DMA-135 оказалась шпилька SL1, расположенная в 5’-UTR, а для DMA-155 –– SL6, которая находится уже в кодирующей последовательности. Ученые планируют произвести дальнейшие модификации выявленных кандидатных препаратов на основе полученных данных, и затем начать КИ на животных.
6.12.2021